De ces études Genome Wide Scan on peut déjà avancer l’idée que certaines maladies multifactorielles auront une base génétique avec un ou des variants génétiques à effets majeurs (comme dans la maladie de Crohn) alors que pour d’autres maladies multifactorielles il sera question d’une multitude de variants génétiques ayant tous un effet mineur (comme cela semble être le cas pour le diabète de type 2 et l’obésité).
Par extension il faut admettre que la dichotomie sémantique entre maladies monogéniques et maladies polygéniques (multifactorielles) est finalement artificielle car entre la mutation d'un gène qui explique toute la variance du trait phénotypique dans une maladie monogénique et une affection multifactorielle dont la composante génétique résulte d'une myriade de variants génétiques à effets faibles il existe un continuum de situations qui expliquent les atteintes de sévérité variable en fonction du background génétique dans des maladies monogéniques et les maladies polygéniques avec l'effet d'un ou plusieurs gènes majeurs.
En effet il est rapporté de nombreux cas de variants génétiques qui modulent la sévérité d'une maladie monogénique comme par exemple dans la mucoviscidose  Drumm ML et al. , Genetic modifiers of lung disease in cystic fibrosis. N Engl J Med 2005 353 pp 1509-1511; Arkwright PD et al. TGF beta 1 genotype and accelerated decline in lung function of patients with cystic fibrosis. Thorax 2000 55 446
Drumm ML et al. , Genetic modifiers of lung disease in cystic fibrosis. N Engl J Med 2005 353 pp 1509-1511; Arkwright PD et al. TGF beta 1 genotype and accelerated decline in lung function of patients with cystic fibrosis. Thorax 2000 55 446
7
.
3
-
L'approche "Genome Wide Association Study" (GWAS)
Le développement des technologies de séquençage et de génotypage à haut débit à partir des années 2000, basées sur les "puces à ADN" (DNA chips) a ouvert la voie à l'obtention rapide d'un très grand nombre de génotypes. Il est alors devenu envisageable de génotyper un grand nombre de marqueurs génétiques chez un grand nombre de sujets. Cette opportunité technologique a ouvert la voie au GWAS qui se résume à une étude d'association à très grande échelle: autant d'analyses d'association cas-témoin que de variants génétiques analysés. En effet la méthode consiste à génotyper un maximum de marqueurs génétiques (de 317 000 à 1 000 000 selon le type de puce à ADN) chez un grand nombre de sujets atteints de la maladie génétique "complexe" et un grand nombre de sujets témoins. Les variations d'un nucléotide (SNP, single nucleotide polymorphism) malgré leur nature bi-allélique qui contraste avec le caractère multi allélique des microsatellites, ont été retenues compte tenu de leur très grand nombre sur le génome humain offrant ainsi une très bonne couverture du génome. En effet les SNPs constituent les variants génétiques les plus fréquents. Par ailleurs leur nature bi allélique les rend plus accessibles au génotypage de masse au moyen des puces à ADN que les marqueurs microsatellites qui présentent un très grand nombre d'allèles. La méthode Genome Wide Scan qui s'intéressait à la co-transmission entre des régions du génome (repérées par les marqueurs microsatellites) et le phénotype "malade" ou "non malade", reposait donc sur la liaison génétique entre les marqueurs microsatellites et les loci en rapport avec la maladie "complexe". La méthode GWAS consiste en des études d'association entre un allèle donné d'un SNP et le phénotype "malade" ou "non malade." La méthode GWAS repose donc sur le déséquilibre de liaison génétique qui existe au sein du génome humain entre un allèle d'un marqueur génétique (SNP) et un variant génétique potentiellement impliqué dans la maladie, variant qui constitue une partie du déterminisme génétique de la maladie "complexe".
L’association mise à profit dans le GWAS est également fonction (1) de la distance entre le locus du marqueur génétique (SNP) analysé et le locus où réside le variant génétique de susceptibilité à la maladie (ce paramètre sera moins limitant compte tenu de la grande densité de marqueurs : un tous les 60kb pour une couverture de 550 000SNPs), (2) mais surtout sera fonction du déséquilibre de liaison génétique (LD pour « linkage disequilibrium ») qui existe entre un allèle donné du locus du marqueur SNP et l’allèle de susceptibilité au niveau du locus où réside le variant génétique de susceptibilité à la maladie. Comme le LD est variable d’une population et d’une ethnie à une autre, les approches GWAS devront prendre en compte cette spécificité et inclure des sujets homogènes quant au LD  Salisbury BA et al. , SNP and haplotype variation in the human genome. Mutat Res. 2003 May 15;526(1-2):53-61
Salisbury BA et al. , SNP and haplotype variation in the human genome. Mutat Res. 2003 May 15;526(1-2):53-61  Evans DM et al., A comparison of linkage disequilibrium patterns and estimated population recombination rates across multiple populations. Am J Hum Genet. 2005 Apr;76(4):681-7. Epub 2005 Feb 17
Evans DM et al., A comparison of linkage disequilibrium patterns and estimated population recombination rates across multiple populations. Am J Hum Genet. 2005 Apr;76(4):681-7. Epub 2005 Feb 17
La méthode GWAS pourrait sembler "la" méthode définitive susceptible de repérer tous les déterminants génétiques d'une malade, compte tenu de sa haute densité de couverture du génome. Néanmoins la méthode souffre d'un certain nombre de limites.
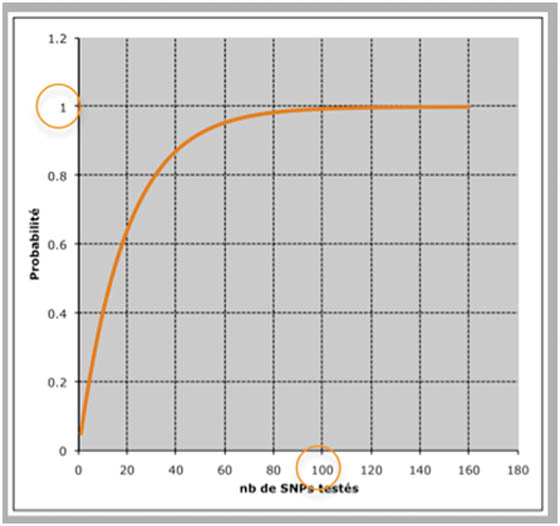
Comme elle se résume à une succession d'études d'association cas-témoin, l'association d'un SNP avec la maladie est testée comme habituellement, au risque statistique 5% (0.05). Une association sera réputée significative quand il n'y a que 5% de chances que cette affirmation soit fausse (ce qui somme toute laisse 95% de chances d'avoir raison). En d'autres termes il y a 5% de chances que l'association soit trouvée positive par hasard. Quand après avoir analysé deux SNPs, on évalue la probabilité que l'un d'entre eux soit associé par hasard, le risque n'est plus de 0.05 mais de 0.1. Il sera de 0.14 pour 3 SNPs....
8/11








 Drumm ML et al. , Genetic modifiers of lung disease in cystic fibrosis. N Engl J Med 2005 353 pp 1509-1511; Arkwright PD et al. TGF beta 1 genotype and accelerated decline in lung function of patients with cystic fibrosis. Thorax 2000 55 446
Drumm ML et al. , Genetic modifiers of lung disease in cystic fibrosis. N Engl J Med 2005 353 pp 1509-1511; Arkwright PD et al. TGF beta 1 genotype and accelerated decline in lung function of patients with cystic fibrosis. Thorax 2000 55 446