7
-
L'identification des facteurs génétiques multiplesimpliqués dans une maladie génétique "complexe"
La recherche et la caractérisation des déterminants génétiques impliqués dans les maladies génétiques "complexes" a été initiée dans la décennie 1990 par des travaux qui ont concerné entre autres le diabète de type 2. Compte tenu de la multiplicité des loci potentiellement impliqués, les méthodes employées dans les maladies monogéniques ont dû être adaptées à l'étude de l'hérédité polygénique. On retiendra de cette époque, deux méthodes majeures: la stratégie gène candidat et l'approche génome entier (Genome Wide Scan).
7
.
1
-
La stratégie gène candidat
Cette stratégie consiste à rechercher et analyser les variants génétiques dans des gènes dont la fonction pourrait jouer un rôle dans la pathologie en question. Elle repose sur une bonne connaissance de la physiologie et de la fonction du gène mis en examen et de ses interactions avec les autres partenaires impliqués dans la même voie métabolique qui pourrait être incriminée dans la pathologie. Par exemple le gène GLUT4 (OMIM138190) codant un transporteur de glucose a été gène candidat dans un contexte de diabète de type 2. Les gènes impliqués dans la réponse immunitaire et l'inflammation ont été candidats dans le cadre du psoriasis. Une fois le ou les variants génétiques identifiés (en général par séquençage direct de l'ADN de sujets atteints et de sujets non atteints) il reste à conclure sur chacun quant à son rôle (ou son absence de rôle) dans la maladie génétique "complexe". Comme dans la maladie "complexe" chaque variant génétique ne possède qu'un effet minime sur la variation du trait phénotypique et donc sur la survenue ou non de la maladie, cette phase nécessite une approche épidémio-génétique sur des groupes importants de patients atteints et de sujets témoins indemnes de la maladie (études cas-témoins).
La question posée étant : le variant génétique est-il plus fréquent chez les sujets présentant la maladie que chez les témoins?
La réponse à cette question ne peut être objectivée que par un test statistique de comparaison de fréquences au risque 5%, dans notre exemple, un test du chi2. Si la réponse est oui, on est en droit de conclure (au risque 5%) que:
- le variant en question est l'un des déterminants génétiques de la maladie
- ou il est en déséquilibre de liaison génétique avec l'un des déterminants génétiques de la maladie.
N'ayant aucune information objective et biologique quant au retentissement du variant génétique sur la fonction du gène et sur la maladie, nous ne pouvons à ce stade que constater qu'il existe une association significative au sens statistique entre la présence du variant génétique et la présence de la maladie. C'est pourquoi ces analyses sont appelées analyses d'association. Ces analyses d'association peuvent aussi se réaliser par des études familiales: tests de déséquilibre de transmission (Transmission disequilibrium test TDT). On objective par un test statistique la réponse à la question : le variant génétique est-il transmis d'un parent hétérozygote à un enfant atteint, plus souvent que ne le voudrait le hasard ?
Ces approches gène candidat qui ont mis en évidence de nombreux variants génétiques impliqués dans des maladies génétiques "complexes", présentent le défaut de ne pouvoir cibler et analyser que les gènes connus dont la fonction est évidemment connue et qui présente un rapport à priori évident avec la pathologie.
7
.
2
-
L'approche génome entier (Genome Wide Scan)
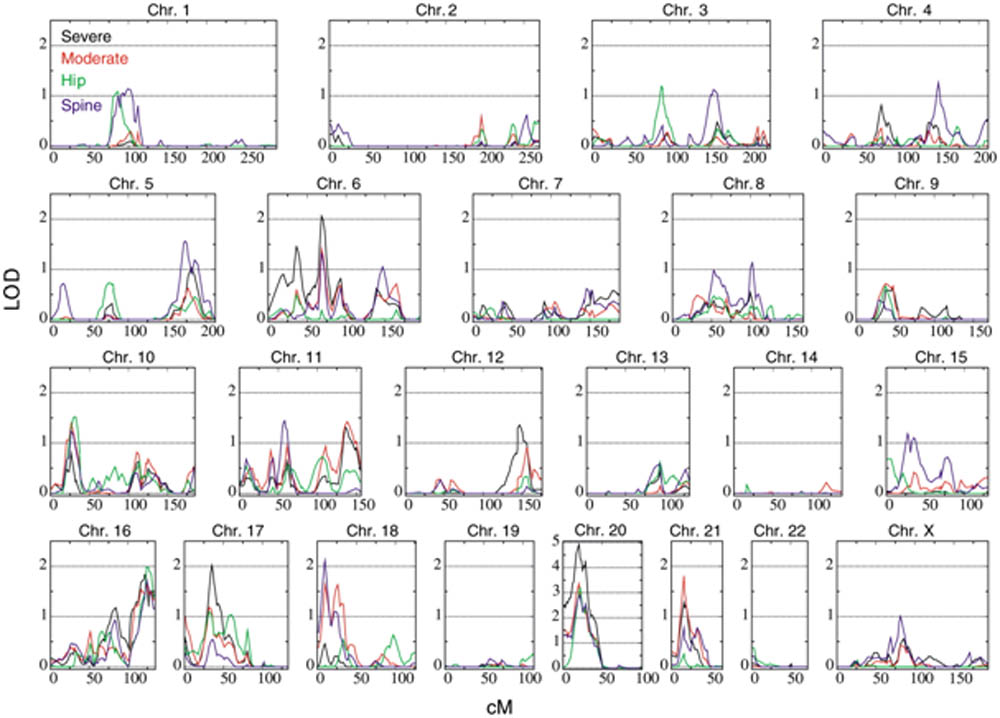
L'approche génome entier consistait à génotyper chez les sujets d'un grand nombre de familles (présentant des patients atteints) un maximum (±1000) de marqueurs microsatellites très polymorphes, recouvrant la totalité du génome. C'est une méthode de génétique inverse et sans à priori contrairement à l'approche gène candidat. L'étude est basée sur l'analyse de la liaison génétique entre chaque marqueur microsatellite et la maladie "complexe". Par analogie avec le calcul du Lodscore employé dans les maladies monogénique et qui quantifie la liaison génétique entre un locus et la maladie, la méthode Genome Wide Scan quantifie la liaison génétique entre chaque marqueur microsatellite et la maladie "complexe". Dans l'exemple ci dessous extrait de Styrkarsdottir U et al.  Styrkarsdottir U et al. , Linkage of osteoporosis to chromosome 20p12 and association to BMP2. PLoS Biol. 2003 Dec;1(3):E69. Epub 2003 Nov 3
Styrkarsdottir U et al. , Linkage of osteoporosis to chromosome 20p12 and association to BMP2. PLoS Biol. 2003 Dec;1(3):E69. Epub 2003 Nov 3
Les résultats montraient clairement que de nombreux loci présentaient une liaison génétique au moins "suggestive" avec l'ostéoporose, en accord avec la nature polygénique de la maladie. Le maximum de liaison génétique était observé sur le chromosome 20.
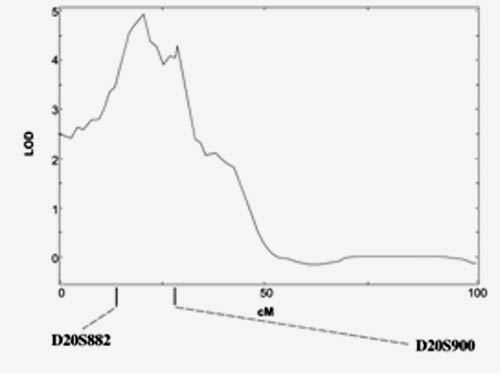
Une vue détaillée des résultats concernant le chromosome 20 montre que la zone de liaison génétique couvrait presque la moitié du chromosome 20: 50cM soit 50 Mégabases d'ADN (50 000 kb). Les investigations ont été restreintes au pic de liaison génétique compris entre les marqueurs D20S882 et D20S900, la zone à explorer à la recherche d'un (ou plusieurs) variant génétique en rapport avec la maladie restait de 1 726 485 bp (1.7Mb) et même si cela ne représentait plus que 3% du chromosome 20 et 0.05% du génome, cette zone à explorer était encore très grande à l'échelle du biologiste moléculaire puisque compte tenu de la densité des gènes sur le chromosome 20 elle était censée contenir en théorie statistiquement 21 gènes  Deloukas P. et al. , The DNA sequence and comparative analysis of human chromosome20. Nature. 2001-27;414(6866):865-71 Il n'est pas rare que la stratégie gène candidat soit utilisée en seconde intention pour restreindre le nombre de gènes dont l'investigation sera être poussée (stratégie dite gène candidat positionnel). Dans l'exemple présenté la région de 1.7 Mb contenait en fait six gène répertoriés et connus en 2003 (BMP2, CHGB, LOC51605, C20orf154, C20orf155, et C20orf42). Compte tenu de son rôle dans la formation de l'os et la différenciation des ostéoblastes le gène BMP2 (Bone Morphogenetic Protein 2 OMIM112261) est apparu le meilleur gène candidat. Des investigations plus fines ont permis de mettre en évidence une mutation Ser37Ala du gène BMP2, fortement associée à l'ostéoporose mais qui n'expliquait que 30% de la liaison génétique sur le chromosome 20.
Deloukas P. et al. , The DNA sequence and comparative analysis of human chromosome20. Nature. 2001-27;414(6866):865-71 Il n'est pas rare que la stratégie gène candidat soit utilisée en seconde intention pour restreindre le nombre de gènes dont l'investigation sera être poussée (stratégie dite gène candidat positionnel). Dans l'exemple présenté la région de 1.7 Mb contenait en fait six gène répertoriés et connus en 2003 (BMP2, CHGB, LOC51605, C20orf154, C20orf155, et C20orf42). Compte tenu de son rôle dans la formation de l'os et la différenciation des ostéoblastes le gène BMP2 (Bone Morphogenetic Protein 2 OMIM112261) est apparu le meilleur gène candidat. Des investigations plus fines ont permis de mettre en évidence une mutation Ser37Ala du gène BMP2, fortement associée à l'ostéoporose mais qui n'expliquait que 30% de la liaison génétique sur le chromosome 20.
On perçoit ici les limites de la méthode qui sont (1) la relative grande taille des régions de liaison génétique (qui n'est pas sans rapport avec la distance entre les microsatellites) et donc le grand nombre de gènes à explorer en détail à la recherche de variants génétiques, (2) la part de liaison restant inexpliquée par les variants génétiques associés à la maladie.
Un dernier exemple incontournable concerne l'approche Genome Wide Scan dans la maladie de Crohn, une maladie inflammatoire chronique de l'intestin qui a conduit à la mise en évidence des mutations du gène NOD2 en cause dans la maladie de Crohn  Hugot JP et al. , Mapping of a susceptibility locus for Crohn's disease on chromosome 16. Nature. 1996 Feb 29;379(6568):821-3
Hugot JP et al. , Mapping of a susceptibility locus for Crohn's disease on chromosome 16. Nature. 1996 Feb 29;379(6568):821-3
La stratégie genome wide scanrepose toujours sur la séquence: analyses de liaison puis clonage positionnel :
Cette mise en évidence du rôle du gène NOD2 représente le modèle type de la stratégie utilisée à l'époque: analyses de liaison puis clonage positionnel :
1°-identification de loci de susceptibilité par analyse de liaison génétique dans des familles,
2°-analyse de liaison avec des marqueurs plus rapprochés dans les loci précédemment identifiés ("fine mapping") pour restreindre la taille du locus de susceptibilité,
3°-puis analyse de gènes ou prédictions de gènes candidats positionnels, dont l'implication dans la maladie est validée ou écartée sur la base d'analyses d'association entre des variants génétiques et la maladie.
7/11







 Styrkarsdottir U et al. , Linkage of osteoporosis to chromosome 20p12 and association to BMP2. PLoS Biol. 2003 Dec;1(3):E69. Epub 2003 Nov 3
Styrkarsdottir U et al. , Linkage of osteoporosis to chromosome 20p12 and association to BMP2. PLoS Biol. 2003 Dec;1(3):E69. Epub 2003 Nov 3