5
-
Consanguinité
Deux personnes sont apparentées lorsqu’ils ont au moins un ancêtre commun vérifiable. Les individus nés d’une union entre apparentés sont dits consanguins. Par extension l’épithète peut concerner une union.
On peut définir le coefficient de consanguinité comme la probabilité que, chez un individu, en un locus autosomique donné, les deux allèles soient identiques par descendance mendélienne c’est-à-dire proviennent de la réplication d’un allèle que possédait un ancêtre commun à son père et à sa mère. L’individu consanguin homozygote à un locus du fait de sa consanguinité peut être dit « autozygote »; le coefficient de consanguinité est donc la probabilité qu’à un locus donné le sujet soit autozygote.
5
.
1
-
Calcul du coefficient
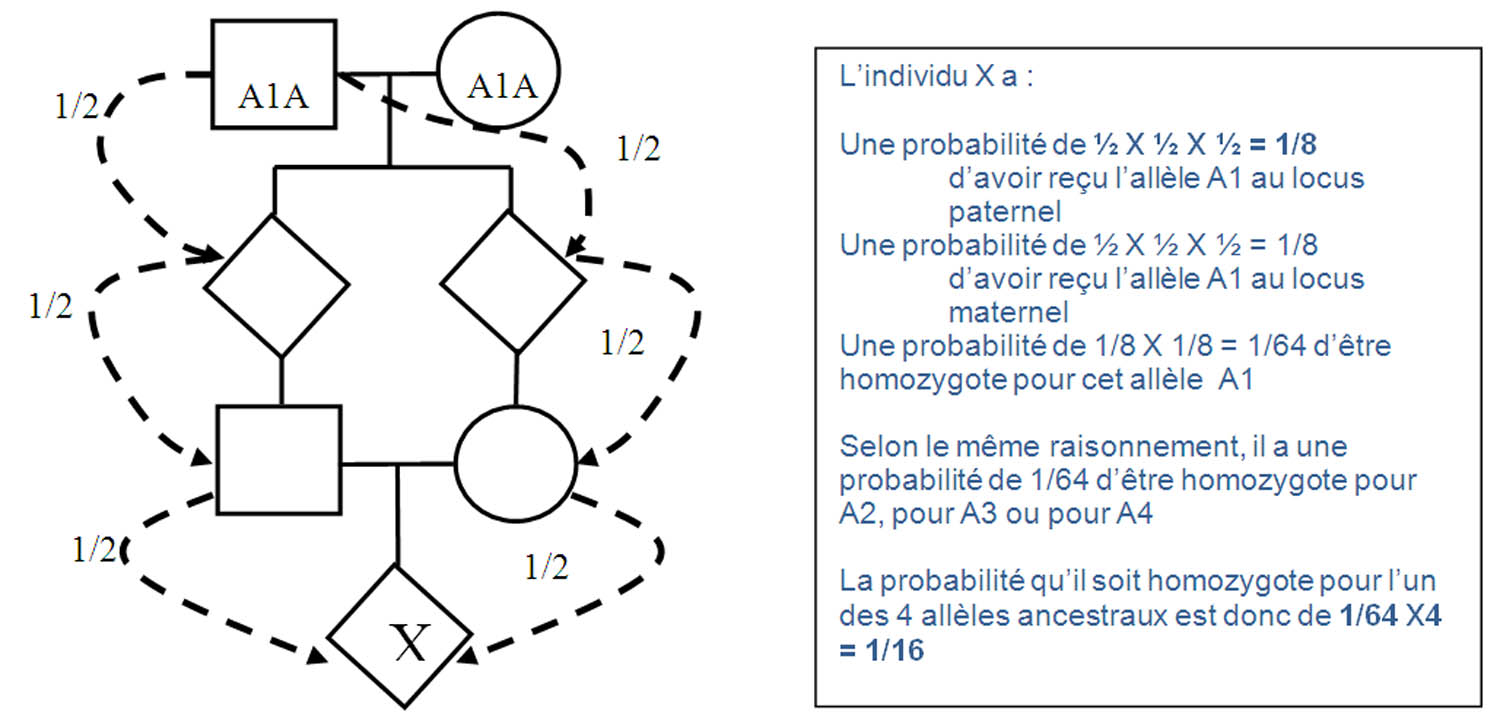
L’individu X, est issu d’un couple de cousins germains :
Soit le locus A. Les deux ancêtres communs sont porteurs respectivement des allèles A1 et A2 et A3 et A4.
Calculons la probabilité que l’individu X soit homozygote au locus A par réplication d’un des ces 4 allèles ancestraux
1/16 = coefficient de consanguinité d’un individu issu de parents cousins germains
Pour chaque allèle, X a (1/2) m+p+2 chances d’être homozygote,
m= nombre de chaînons qui relient la mère à l’ancêtre commun
p= nombre de chaînons qui relient le père à l’ancêtre commun
Pour chaque ancêtre, X a 1/2 (m+p+1) (= 2x 1/2 (m+p+2) ) chances d’être homozygote.
Coefficient de consanguinité F F = Σ 1/2 (m+p+1)
∑ = somme (on calcule 1/2 (m+p+1) pour chaque ancêtre)
5
.
2
-
Consanguinité et maladie récessive autosomique
A l’échelle d’un individu
Soit une maladie de fréquence q2 (un individu pris au hasard dans la population générale a un risque q2 d’être atteint de cette maladie).
Pour un individu consanguin (coefficient F) :
- La probabilité que cet individu soit homozygote du fait de sa consanguinité est Fq.
- La probabilité qu’il soit homozygote du fait du hasard et de (1-F)q2
- Le risque qu'il soit homozygote, au total est
Fq +(1-F)q2
soit = q2 - F q2 + Fq
soit = q2 + Fq (1-q)
soit = q2 + Fq (car 1-q = p, et p est très proche de 1)
La probabilité d'avoir un enfant homozygote pour un allèle muté, donc atteint, dans le cas d’une maladie autosomique récessive dont la fréquence est q2 , dans un mariage consanguin où le coefficient de parenté est F, est égal à q2+ Fq
On trouve une plus grande proportion d’individus apparentés parmi les parents d’enfants atteints de maladies récessives.
Risque relatif d'avoir un enfant atteint d’une maladie autosomique récessive (de fréquence q2) dans un couple consanguin par rapport à la population générale
Fq + (1-F)q2
RR = ------------------------
q2
On montre que plus la maladie est rare, plus le risque relatif pour les unions consanguines est important
Exemple : risque d’avoir un enfant atteint de Phénylcétonurie pour un couple de cousins germains.
Fréquence de la phénylcétonurie dans la population générale : q2 = 1/10000
R (risque lié à la consanguinité) = 1 / 10000 + (1/16 x 1/100)
= 1 / 10000 + 1/1600
= 1/1600
RR = risque lié à la consanguinité / risque de la population générale
1/1600 / 1/10 000 = 6
Le risque est 6 fois plus élevé pour des cousins germains que pour un couple non apparenté.
Le risque est d’autant plus élevé que la maladie est rare : Exemples:
Mucoviscidose (q2 = 1/3000) : RR =3.5
Galactosémie (q2 = 1/40000) : RR =12
Ataxie télangiectasie (q2 = 1/100000) : RR =20
5
.
3
-
Les conséquences de la consanguinité à l’échelle d’une population
Dans des populations fortement consanguines, quel est le retentissement sur les fréquences alléliques et génotypiques?
On peut définir Fi = coefficient moyen de parenté dans une population non panmictique
Fi = moyenne des coefficients de parenté des différents couples de la génération i
Soit un locus di-allélique : allèles A1 et A2, de fréquences respectives p et q chez les parents. Soit F le coefficient moyen de parenté à la génération des parents.
A la génération suivante :
f(A1A1) = Fp + (1-F)p2
f(A2A2) = Fq + (1-F)q2
f(A1A2) = Fpq + (1-F)2pq
Soit:
f(A1A1) = p2 + Fp (1-p)
f(A2A2) = q2 + Fq (1-q)
f(A1A2) = 2pq – 2Fpq
Il y a donc un accroissement de la fréquence des homozygotes, et une diminution de la fréquence des hétérozygotes. (fréquences génotypiques)
En revanche, les fréquences alléliques restent inchangées
Ex: f(A1) = (p2 + Fp (1-p)) + (1/2 (2pq – 2Fpq)) = p2 + Fpq + pq –Fpq = p2 + pq = p (p+q) = p
Donc: la consanguinité ne s’accompagne pas de détérioration du stock génique dans une grande population, mais d’une augmentation de la morbidité pour les maladies autosomiques récessives.

6/6