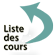1
.
2
-
L’hybridation génomique comparative sur réseau d'ADN ou CGH Array
1
.
2
.
1
-
Un peu d’histoire
En 1992, l’équipe de Dan Pinkel mit au point une nouvelle technique de cytogénétique appelée hybridation génomique comparative (CGH) (6). Le principe est de comparer le nombre de molécules d’une même quantité d’ADN de deux individus en les marquant chacune par un fluorochrome différent puis en les hybridant ensemble sur des métaphases d’un témoin. Après l’étape d’hybridation, les signaux générés par les deux fluorochromes sont numérisés et un rapport de leur intensité respective – patient sur témoin (reflétant le rapport du nombre de molécule d’ADN du patient par rapport à celle du témoin) - est établi au niveau des bandes de chacune des paires chromosomiques. Le ratio d’intensité des deux fluorochromes au niveau d’une bande aura une valeur théorique proche de 1 si le nombre de molécules des deux génomes est identique au niveau de la bande observée. En cas de délétion, ce rapport théorique sera de 0,5 et de 1,5 en cas de trisomie (figure 5). Une des limites de la CGH sur métaphases, est de ne détecter que des déséquilibres génomiques. Les anomalies chromosomiques équilibrées et les anomalies en faible mosaïque ne sont pas mis en évidence.
Ainsi, cette technique a permis de s’extraire de l’analyse morphologique des chromosomes d’un patient en analysant son contenu global en ADN. Mais la résolution de la CGH sur métaphases reste celle de la bande chromosomique (5–10 Mb), car le ratio d’intensité des fluorochromes est établi au niveau de chaque bande chromosomique.
Vers la fin des années 1990, Solinas-Toldo (7) et Pinkel et al. (8) proposent une autre technique de CGH sur des lames sur lesquelles sont fixés des fragments d’ADN. Il s’agit de la CGH sur réseau d'ADN ou CGH array !
1
.
2
.
2
-
Principe
À la fin des années 90, ces auteurs démontrent qu’il est possible d’effectuer une CGH sur des fragments d’ADN fixés sur des lames de verre. Très rapidement, avec le séquençage du génome, il fut possible de fixer sur ces lames, des séquences génomiques, appelées « sondes », représentant une partie plus ou moins importante du génome. Ces lames furent appelées « puce à ADN » ou microarray. Comme pour la CGH sur métaphases, une même quantité d’ADN témoin et d’ADN patient marquée par deux fluorochromes différents (ce mélange est appelé « cible ») sont déposées sur la lame. Le rapport d’intensité de fluorescence est calculé au niveau de chaque fragment d’ADN fixé (appelé « sonde »). Un traitement statistique des données est ensuite réalisé grâce à des logiciels dédiés. Les résultats sont donnés sous forme graphique où un « point » correspond à la valeur du ratio d’intensité de fluorescence au niveau d’une sonde (figure 6). L’ensemble des « points » est placé sur un idéogramme pour chaque chromosome. À la différence de la CGH sur métaphases, l’existence d’un déséquilibre génomique sera objectivée par une déviation du ratio d’intensité de fluorescence non plus au niveau d’une bande chromosomique, mais au niveau des sondes fixées sur la lame. De leur nombre et de leur localisation sur le génome dépendra la résolution de la puce utilisée.
Les premières « puces pangénomiques » (représentant l’ensemble du génome), furent construites avec 3000 clones dont la localisation correspondait à des loci espacés toutes les mégabases. Le génome étant d’une taille de 3 milliards de paires de base, ces puces avait une résolution moyenne d’une mégabase, sois 5 fois celle d’un caryotype en haute résolution. Ishkanian et al. (9) en utilisant 32 433 BACs chevauchants sur tout le génome ont pu obtenir une puce avec une résolution de l’ordre de 30 kb.
Depuis le milieu des années 2000, les puces à oligonucléotides firent leur apparition. Il s’agit de lames sur lesquelles sont fixés des oligonucléotides d’environ 60-80 bases (ou « mer ») qui peuvent être déposés ou directement synthétisés dessus. Le nombre d'oligonucléotides peut atteindre plusieurs millions ce qui permet d'obtenir une résolution d’environ mille paires de base. Ces lames sont aujourd’hui commercialisées par de nombreuses compagnies.
La CGH array permet la détection de déséquilibres chromosomiques dont la taille varie avec la résolution de la puce utilisée. Parce que la CGH est une comparaison de nombre de molécules d’ADN d’un individu par rapport à un autre, un déséquilibre génomique de plus de 1 kb détecté par cette technique a été appelé CNV pour « Copy Number Variant » ou en français « Variation de Nombre de Copies ».
1
.
2
.
3
-
Une variante : les puces à SNPs (figure 7)
ll est possible de fixer sur les lames, des oligonucléotides contenant des « Single Nucleotide Polymorphims » ou SNPs. Ce sont des variations portant sur une seule paire de base, et distribuées uniformément dans le génome humain. On estime le nombre de SNPs à plus de 10 millions avec une répartition d’un SNP tous les 100 à 1000 paires de bases (1kb). La carte des SNPs étant établie, différents procédés ont été mis au point pour synthétiser des oligonucléotides contenant pour un locus donné les 2 allèles du SNP. L’avantage de l’utilisation de ces puces dites puces à SNPs est de pouvoir détecter les situations d’isodisomies uniparentales (perte d’hérérozygotie) et de déterminer l’origine parentale d’un remaniement (avec l’étude des parents). Pour la plupart des sondes, l’hybridation s’effectuera sur les deux allèles. En cas de perte d’hétérozygotie, un seul allèle sera détecté. Celle-ci aura une signification lorsqu’elle impliquera plusieurs sondes adjacentes.
Ces puces ont initialement été développées à des fins d’haplotypage et d’analyse de liaison. Cependant, elles donnent également une information sur le nombre de copies d’ADN aux loci étudiés.
Ces deux techniques sont aujourd’hui totalement automatisables et permettent de ce fait l’étude des déséquilibres génomiques à haut débit. L’acronyme ACPA qui signifie « Analyse Chromosomique sur Puces à ADN » (acronyme choisi par le réseau français AChro-Puce, http://www.renapa.univ-montp1.fr/) désigne aussi bien la technique de CGH array que celle des puces à SNPs.


2/5