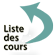Introduction
La cytogénétique médicale est en pleine révolution. Le caryotype mis au point en 1956, n’est désormais plus l’examen de première intention pour l’exploration des anomalies chromosomiques associées à une déficience intellectuelle et/ou aux malformations congénitales (DI et/ou MC). Il est aujourd’hui supplanté par l’hybridation génomique sur réseau d’ADN ou CGH array qui est l’aboutissement du développement des techniques de cytogénétique moléculaire dont le début remonte aux années 80. La CGH array détecte des déséquilibres génomiques avec une résolution 10 à 500 fois supérieure à celle du caryotype (qui est de 5 à 10 millions de paires de bases ou 5-10 Mb). De ce fait, la taille d’un segment chromosomique remanié peut être déterminée avec précision ce qui permet de connaître son contenu génique et donc de faciliter l’établissement des corrélations phénotype – génotype. La détection d’une perte ou d’un gain de matériel chromosomique chez des patients présentant une DI et/ou MC mais aussi chez des patients avec des pathologies psychiatriques ou neurologiques permet dans certains cas d’identifier le(s) gène(s) impliqué(s). Avec l’avènement de cette technique, le cytogénéticien « constitutionnaliste » devient un « interniste» de la pathologie génomique.
Dans les cancers et en particulier dans les hémopathies malignes, l’impact de la CGH array, pour le diagnostic des anomalies chromosomiques est plus limité. En effet, les anomalies chromosomiques qui leurs sont associées, sont d’une part souvent équilibrées (la CGH ne détecte que les déséquilibres génomiques) et seules certaines d'entre elles sont recherchées pour le traitement. De ce fait, la FISH (qui est une technique de cytogénétique ciblée) et la RT-PCR restent les examens moléculaires de choix, avec le caryotype, pour le diagnostic en routine des anomalies présentes dans ces pathologies.
Par ailleurs, sur le plan de la recherche, la CGH array est un outil précieux pour l’identification de gènes impliqués dans des pathologies constitutionnelles et acquises.
Dans ce chapitre, nous exposerons dans un premier temps les bases techniques des principaux outils de la cytogénétique moléculaire utilisés en routine dans les laboratoires de cytogénétique puis nous présenterons les principales applications de ces techniques. Enfin, nous terminerons sur une réflexion concernant l’avenir de la cytogénétique.
1
-
Cytogénétique moléculaire, aspects techniques
1
.
1
-
L’hybridation in situ fluorescente ou FISH (Fluorescence in situ hybridization)
1
.
1
.
1
-
Un peu d’histoire
La toute première technique de cytogénétique moléculaire s’appelle l’hybridation in situ fluorescente. Elle repose sur les propriétés de dénaturation et de renaturation de la molécule d’ADN. Dans certaines conditions de température, de ph ou de salinité, les deux brins d’une molécule d’ADN peuvent se séparer (phénomène appelé dénaturation) puis se réassocier de façon spécifique (étape appelée la renaturation).
En 1981, l’équipe de David Ward intègre par voie chimique un nucléotide (du dUTP) couplé à de la biotine dans un fragment d’ADN (cette opération s’appelle le « marquage ») qui est alors appelé « sonde ». Dénaturée, puis hybridée sur des préparations chromosomiques, elles aussi préalablement dénaturées, la sonde est révélée par des anticorps anti-biotine couplés à un fluorochrome, le FITC. Grâce à un microscope qui émet un faisceau lumineux excitant le FITC, David Ward visualisa directement sur des chromosomes la localisation de la sonde. Ainsi naquit l’hybridation in situ fluorescente, permettant l’observation de loci sur des métaphases ou des noyaux, d’ou le terme in situ (à la différence du Southern blot qui est l’hybridation d’une sonde sur de l’ADN fixé sur une membrane de nylon) (1). L’utilisation de plusieurs fluorochromes et de filtres microscopiques idoines ainsi que le développement de système de numérisation des signaux fluorescents ont permis d’hybrider plusieurs sondes de façon concomitante. Grâce à ces progrès, il est possible aujourd’hui d’étudier de façon simultanée plus de 20 loci sur des chromosomes.
1
.
1
.
2
-
Les sondes
Avec les progrès des techniques, il est possible de générer aujourd’hui des fragments d’ADN de tailles variées correspondant à différentes parties d’un chromosome. Pour la FISH, on utilise des sondes spécifiques de régions chromosomiques ou des sondes capables de s’hybrider sur les bras d’une paire chromosomique donnée.
On distingue :
-
Les sondes composées de séquences spécifiques d’ADN répété. Elles sont de petite taille (moins de 1000 paires de bases ou 1 kilobase), mais s’hybrident sur des séquences spécifiques (centromères par exemple) répétées en tandem sur plusieurs centaines de kilobases. Elles génèrent des signaux ponctuels de forte intensité (figure 1a).
-
Les sondes composées de séquences uniques. On distingue les sondes spécifiques de loci et les sondes spécifiques d’un bras chromosomique ou d’un chromosome entier (figure 1b et 1c).
-
Les sondes spécifiques de loci : Pour être entièrement séquencé, le génome humain fut fragmenté en segments de 100-200 kb puis clonés dans des vecteurs appelés BACs (« chromosomes artificiels de bactéries »). Ces BACs sont aujourd’hui utilisés comme sondes pour la technique de FISH. Le choix du BAC se fait par l’intermédiaire de sites internet publics donnant leur position précise sur le génome humain (UCSC genome.ucsc.edu/ -, Ensembl www.ensembl.org/ -, Database of Genomic Variants projects.tcag.ca/variation). D’autres vecteurs peuvent être également utilisés comme sondes (fosmides, cosmides, phages, plasmides ou chromosomes artificiels de levure ou YACs).
-
Les sondes de peinture chromosomique : Par PCR à partir d’ADN de lignées d’hybrides somatiques contenant chacune un exemplaire d’une paire de chromosome humain ou de chromosomes obtenus par cytométrie de flux, il est possible d’obtenir un ensemble de fragments d’ADN représentant un chromosome en entier. Ces fragments marqués vont s’hybrider sur une paire chromosomique donnée. L'ensemble de ces fragments s'appelle « sonde de peinture chromosomique » (2).
1
.
1
.
3
-
Le marquage
1
.
1
.
3
.
1
-
Principe général
Le marquage est l’étape qui permet d’introduire des fluorochromes dans un fragment d’ADN. Au début des années 80, on utilisait des nucléotides, le plus souvent le dUTP, couplé aux haptènes que sont la digoxigénine et la biotine. Les sondes étaient alors révélées grâce à des anticorps antidigoxigénine ou de la streptavidine (substance se fixant spécifiquement sur la biotine) couplés à des fluorochromes. Aujourd’hui, les fluorochromes sont directement fixés sur les nucléotides. Différents procédés sont utilisés pour incorporer un fluorochrome dans un fragment d’ADN. Les plus connues sont le Random-priming et la Nick-translation.
1
.
1
.
3
.
2
-
Les fluorochromes
Les fluorochromes sont des molécules capables d’être excitées (accumulation d’énergie) par une longueur d’onde donnée, appelée longueur d’onde d’excitation (λexc) et de restituer une partie de cette énergie sous l’aspect d’une longueur d’onde de moindre énergie appelée longueur d’onde d’émission (λem). Ils sont donc tous caractérisés par une longueur d’onde d’excitation et une longueur d’onde d’émission.
Les fluorochromes couramment utilisés sont :
- le DAPI (4’,6-diamidino-2-phenylindole) (310nm – 372nm) se fixe sur les régions riches en AT de l’ADN et est appliqué directement sur les préparations chromosomiques après l’hybridation pour colorer de façon aspécifique tous les chromosomes, ce qui permet de les repérer.
- le FITC (Fluorescein isothiocyanate) (exc 495nm – λem 519nm) fluoresce dans le vert
- La Cyanine 3 (Cy3) (λexc 495nm – λem 519nm) fluoresce dans l’orange
- Le Texas red (λexc 589nm – λem 615nm) fluoresce dans le rouge
- La Cyanine 5 (λexc 650nm – λem670nm) fluoresce dans le rouge
- La Cyanine 5.5 (λexc 675nm – λem 694nm) fluoresce dans le rouge
1
.
1
.
3
.
3
-
La multifluorescence
La FISH offre la possibilité d'étudier plusieurs loci simultanément lors d’une seule hybridation. Il suffit pour cela d'hybrider des sondes marquées avec des fluorochromes différents dont les spectres d'excitation et d'émission ne se chevauchent pas.
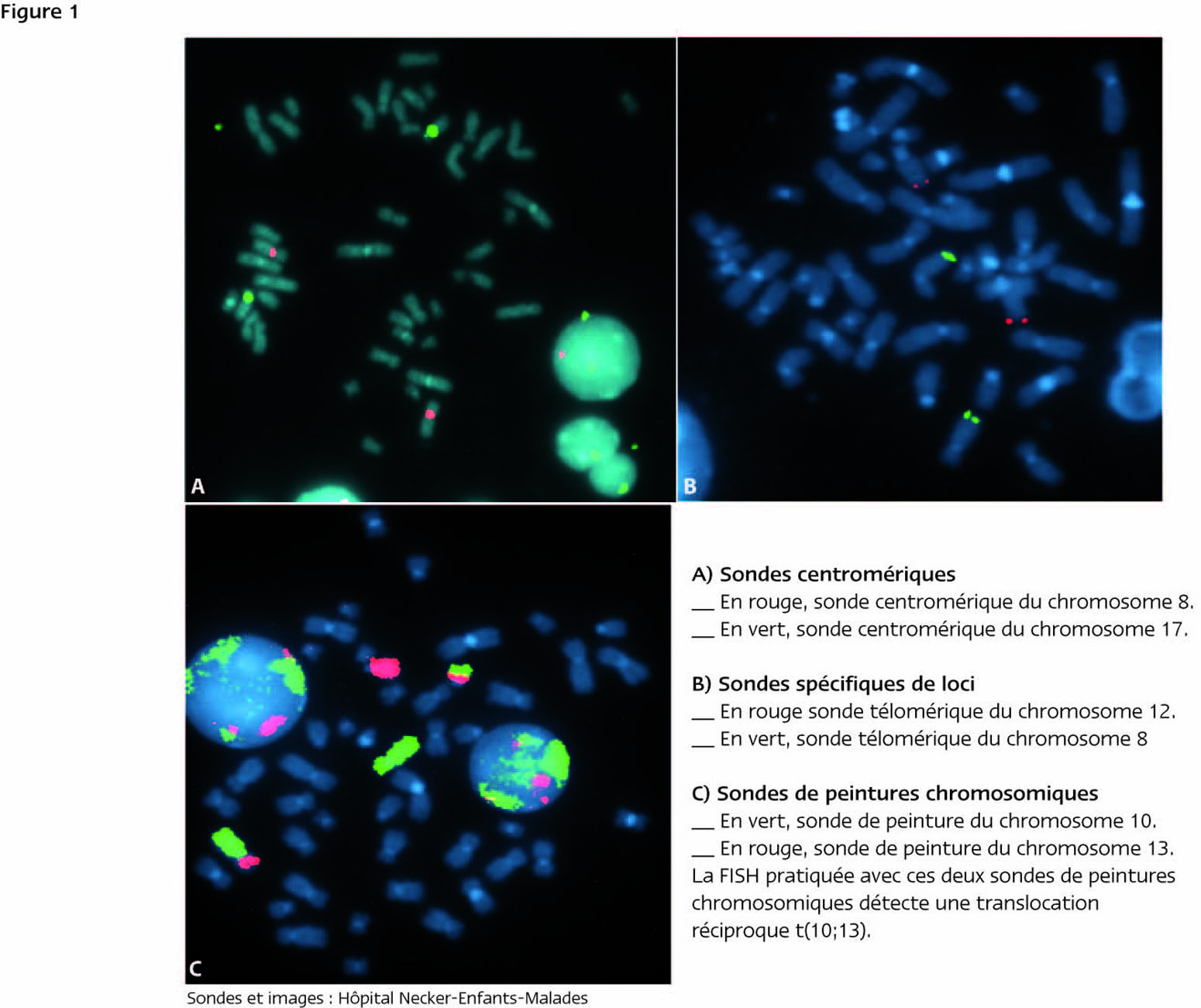
Il est également possible de marquer une sonde avec plusieurs fluorochromes. On obtient alors une sonde dont la fluorescence est complexe, composée des longueurs d'émission des différents fluorochromes. Le signal de ce type de sonde ne peut être analysé qu'après numérisation (voir paragraphe e.i). En utilisant 5 fluorochromes (avec lesquels 31 combinaisons sont possibles), il est possible de construire 23 sondes de peinture, chacune spécifique d'une paire chromosomique donnée, ouvrant la voie au caryotype en multifluorescence (figure2a). Par le même procédé, on peut obtenir des sondes spécifiques de chaque extrémité subtélomérique ou de chaque centromère (figure2b et 2c).
a) L'hybridation (figure 3)
C'est l'opération qui consiste à mettre en présence la sonde dénaturée généralement par la chaleur et l'ADN des chromosomes et des noyaux également dénaturés. Son efficacité dépend du temps d'hybridation et de la concentration de la sonde. Le temps d'hybridation varie de 5 minutes pour des sondes centromériques, à 24 ou 48 heures pour les sondes plus complexes comme les sondes de peinture.
Dans la grande majorité des cas, l'hybridation est dite « compétitive ». En effet, l'ADN est composé de 40% de séquences répétées et de 65% de séquences uniques, ce qui correspond à la composition des sondes (à l’exception des sondes centromériques et télomériques). Après marquage, ces séquences répétées vont s’hybrider sur leurs cibles génomiques engendrant des signaux aspécifiques. Pour les éviter, Lengoer et al. en 1986 ont eu l'idée d'ajouter à l’ADN de la sonde un excès d'ADN non marqué riche en séquences répétées (appelé aujourd'hui ADN Cot1). Ainsi cet ADN s’hybride non seulement sur les séquences répétées de la sonde mais aussi sur celles des cibles génomiques. Cette méthode appelée hybridation « compétitive » permet d'éviter les signaux aspécifiques (3).
b) La microscopie en épifluorescence
Principe général
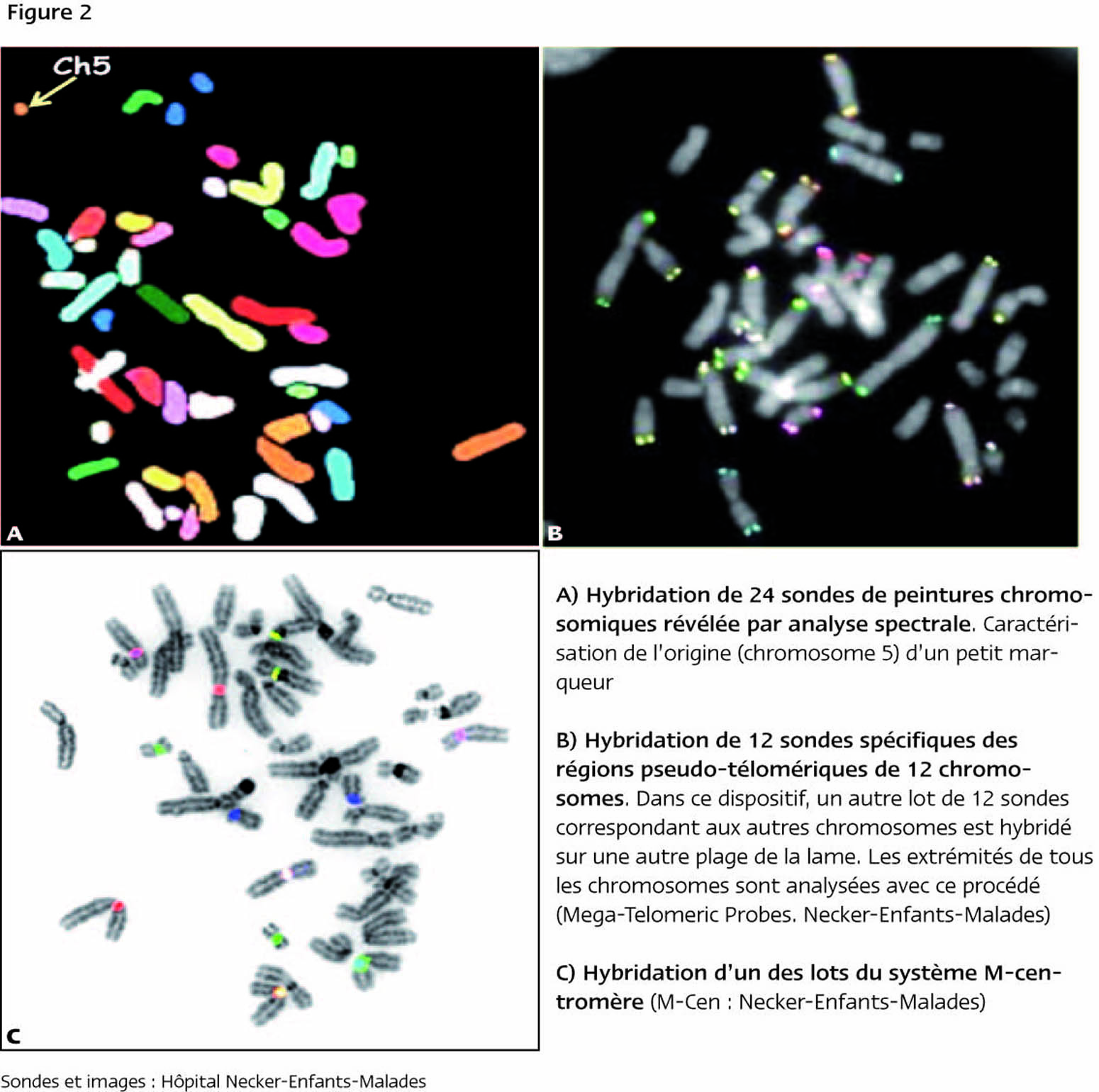
La figure 4 illustre la microscopie en épifluorescence. Un rayon lumineux de lumière blanche (contenant l’ensemble des longueurs d’onde de l’ultra violet aux infrarouges) est généré par des lampes à mercure (donnant des pics d’intensité dans les longueurs d’onde du visible) ou à Xénon (pics d’intensité homogène pour toutes les longueurs d’onde).
Cette lumière est transmise au niveau d’un dispositif contenant deux filtres et un miroir dichroïque. Le premier filtre sélectionne une longueur d’onde d’excitation (correspondant au fluorochrome intégré dans la sonde d’intérêt) qui est ensuite réfléchie par le miroir dichroïque sur la lame hybridée. Une fois le fluorochrome excité, il émet une longueur d’onde d’émission, d’intensité plus faible, qui peut alors passer à travers le miroir dichroïque pour arriver ensuite au filtre d’émission de façon à éliminer des longueurs d’onde parasites. Cette longueur d’onde d’émission est par la suite transmise aux oculaires du microscope pour être observée par l’œil de l’observateur ou au niveau d’une caméra qui numérise ce signal. En changeant de filtres, on peut alors étudier de cette façon plusieurs sondes marquées par des fluorochromes différents.
La multifluorescence
La possibilité de marquer des sondes avec plusieurs fluorochromes, le développement des caméras permettant la numérisation des signaux et la mise au point de différents logiciels d’analyse des signaux, ont permis le développement du caryotype en multifluorescence. Deux procédés sont utilisés :
Le premier repose sur la numérisation successive par la caméra des différents signaux émis par les sondes hybridées. Souvent, la première image capturée est celle générée par le DAPI. La superposition de l’ensemble des signaux sur un écran d’ordinateurs permettra d’étudier précisément les loci correspondant aux sondes hybridées. En utilisant 23 sondes de peinture avec un logiciel ayant intégré les combinaisons des fluorochromes spécifiques de chaque paire chromosomiques, Speicher et al (1996) ont été capables d’établir un caryotype en multifluorescence en classant des chromosomes d'après leur fluorescence spécifique (4).
Le deuxième procédé repose sur l’analyse spectrale des signaux. C’est une méthode d’analyse des signaux plus complexe qui nécessite un interféromètre dont le rôle est de décomposer et d’analyser, en un seul temps, le contenu en longueurs d’onde d’une lumière complexe. Ce procédé fut décrit en 1996 par l’équipe de Schrock et al (5)
c) La limite de la technique de FISCH
La FISH avec des sondes spécifiques de loci est une technique d’étude ciblée, à la différence du caryotype qui permet d’explorer l’ensemble du génome. La FISH avec les sondes de peinture a une résolution de 1,5Mb pour la détection des translocations télomériques. En cas d’insertion chromosomique, elles ne permettent pas de déterminer la région chromosomique impliquée. Enfin, elle ne sont pas d’une grande utilité pour la détection des duplications.

1/5