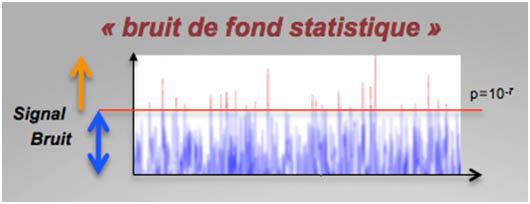
On voit qu'à partir d'une centaine de SNPs testés la probabilité que l'un d'entre eux soit associé à la maladie par hasard (calculée par la loi binomiale) est quasiment de 1. Une méthode classique pour prendre en compte ce problème est de diminuer le seuil de significativité individuel de 0.05 à 0.025 dans le cas de deux SNPs. C'est le principe de la correction de Bonferoni. Mais à combien faut-il baisser le seuil de significativité individuel de chaque SNP pour que globalement une fois les 550 000 SNPs testés, le risque que l'un d'entre eux soit positif par hasard soit encore ≤ 0.05 ? Le calcul montre que si on teste chaque SNP au risque 0.0000001 (10-7), la probabilité pour que l'un d'entre eux soir déclaré associé à la maladie par hasard est voisine de 0.05, ce qui devient statistiquement acceptable.
Il apparaît donc que seuls les SNPs présentant une association très fortement significative (p≤10-7), pourront être considérés comme étant très vraisemblablement associés à la maladie et donc physiquement proches d'un déterminant génétique de la maladie. Une proximité qui sera fonction de la densité des marqueurs génétiques. La méthode GWAS aura donc tendance à ne pouvoir détecter que les effets génétiques importants. C'est un phénomène que l'on peut présenter comme un "bruit de fond statistique".
Une manière de contourner ce problème est de travailler sur des effectifs pléthoriques de patients et de témoins. En effet pour un même effet biologique ou physiologique testé, si la probabilité associée à la significativité du test statistique est de 0.05 avec 100 patients et 100 témoins elle peut être de 0.001 avec deux fois plus de sujets. C'est l'une des raisons pour laquelle il n'est pas rare que des protocoles GWAS incluent un grand nombre de sujets.
La méthode GWAS reste néanmoins une étude d'association qui implique qu'une détection ne sera possible seulement que si le déterminant génétique est en déséquilibre de liaison génétique avec le marqueur SNP génotypé. Par ailleurs les SNPs inclus dans les puces à ADN par les fabricants, ont été choisis parmi les SNPs « fréquents » en terme de fréquences alléliques pour des raisons de commodité technologique et d'ubiquité d'utilisation. La méthode repose donc implicitement sur le postulat « common disease - common variant » qui s'avère de moins en moins ubiquitaire  Pritchard JK, Cox NJ, The allelic architecture of human disease genes: common disease-common variant...or not? Hum Mol Genet. 2002 Oct 1;11(20):2417-23. L’hypothèse « common disease common variant » suppose que les maladies multifactorielles qui sont des maladies fréquentes, reposeraient sur une susceptibilité génétique résultant de variants génétiques fréquents en termes de fréquences alléliques. La méthode GWAS présente donc la tendance à faire l'impasse sur les variants génétiques rares. Cependant il est manifeste que certains variants génétiques rares jouent un rôle non négligeable de la susceptibilité génétique dans les maladies multifactorielles. Par exemple pour la mutation Y111H du gène ADIPOQ la fréquence allélique du variant n'est que de 0.015 (variant rare) mais cette variation s'accompagne d'un sur-risque de diabète de type 2 avec un OR=7.85
Pritchard JK, Cox NJ, The allelic architecture of human disease genes: common disease-common variant...or not? Hum Mol Genet. 2002 Oct 1;11(20):2417-23. L’hypothèse « common disease common variant » suppose que les maladies multifactorielles qui sont des maladies fréquentes, reposeraient sur une susceptibilité génétique résultant de variants génétiques fréquents en termes de fréquences alléliques. La méthode GWAS présente donc la tendance à faire l'impasse sur les variants génétiques rares. Cependant il est manifeste que certains variants génétiques rares jouent un rôle non négligeable de la susceptibilité génétique dans les maladies multifactorielles. Par exemple pour la mutation Y111H du gène ADIPOQ la fréquence allélique du variant n'est que de 0.015 (variant rare) mais cette variation s'accompagne d'un sur-risque de diabète de type 2 avec un OR=7.85  Vasseur F et al. , Single-nucleotide polymorphism haplotypes in the both proximal promoter and exon 3 of the APM1 gene modulate adipocyte-secreted adiponectin hormone levels and contribute to the genetic risk for type 2 diabetes in French Caucasians. Hum Mol Genet. 2002 Oct 1;11(21):2607-14
Vasseur F et al. , Single-nucleotide polymorphism haplotypes in the both proximal promoter and exon 3 of the APM1 gene modulate adipocyte-secreted adiponectin hormone levels and contribute to the genetic risk for type 2 diabetes in French Caucasians. Hum Mol Genet. 2002 Oct 1;11(21):2607-14
Néanmoins la méthode GWAS a permis la mise en évidence de nombreux nouveaux variants génétiques de susceptibilité pour toutes les maladies multifactorielles et les divers QTLs analysés. Evidemment la méthode a retrouvé les associations antérieurement rapportées avec les méthodes antérieures comme par exemple les mutations du gène NOD2 dans la maladie de Crohn, les variants du gène APOE dans la maladie d’Alzheimer, les variants du gène PPARG dans le diabète de type 2. Une revue récente de 2010, répertoriait 49 gènes de susceptibilité au diabète de type 2, la plupart mis en évidence par GWAS  Billings LK, Florez JC. , The genetics of type 2 diabetes: what have we learned from GWAS? Ann N Y Acad Sci. 2010 Nov;1212:59-77. doi: 10.1111/j.1749-6632.2010.05838.x De même une étude de 2010 rapportait 71 loci de susceptibilité à la maladie de Crohn la plupart détectés au moyen de la méthode GWAS
Billings LK, Florez JC. , The genetics of type 2 diabetes: what have we learned from GWAS? Ann N Y Acad Sci. 2010 Nov;1212:59-77. doi: 10.1111/j.1749-6632.2010.05838.x De même une étude de 2010 rapportait 71 loci de susceptibilité à la maladie de Crohn la plupart détectés au moyen de la méthode GWAS  Franke et al. , Genome-wide meta-analysis increases to 71 the number of confirmed Crohn's disease susceptibility loci. Nat Genet. 2010 Dec;42(12):1118-25 On voit que la méthode a permis une avancée significative dans la mise en évidence des déterminants génétiques en cause dans les maladies multifactorielles.
Franke et al. , Genome-wide meta-analysis increases to 71 the number of confirmed Crohn's disease susceptibility loci. Nat Genet. 2010 Dec;42(12):1118-25 On voit que la méthode a permis une avancée significative dans la mise en évidence des déterminants génétiques en cause dans les maladies multifactorielles.
En savoir plus: Why do genome-wide scans fail? http://www.genetic-future.com/2008/03/why-do-genome-wide-scans-fail.html;  Lango H Weedon MN , What will whole genome searches for susceptibility genes for common complex disease offer to clinical practice? J Intern Med 2008 vol. 263 (1) pp. 16-27. Manolio TA Genomewide association studies and assessment of the risk of disease N Engl J Med 2010 vol. 363 (2) pp. 166-76.
Lango H Weedon MN , What will whole genome searches for susceptibility genes for common complex disease offer to clinical practice? J Intern Med 2008 vol. 263 (1) pp. 16-27. Manolio TA Genomewide association studies and assessment of the risk of disease N Engl J Med 2010 vol. 363 (2) pp. 166-76.
7
.
4
-
Le "Whole Exome Sequencing"
Malgré les avancées spectaculaires dans la connaissance des facteurs génétiques de susceptibilité aux maladies multifactorielles réalisées entre autres via les GWAS, pour une maladie donnée, l’ensemble des variants identifiés n’explique qu’une faible partie de la variance du phénotype (héritabilité). En moyenne dans les maladies multifactorielles à peine 10% de l’héritabilité est expliquée par les variants génétiques connus. La question est alors « sur quoi reposent les 90% manquants ? » et quelles méthodes d’investigation employer pour caractériser cette part manquante et ainsi expliquer 100% de l’héritabilité?
Une approche récente du problème repose sur une opportunité technologique. Partant du principe que les séquences codantes ne représentent qu'une faible partie du génome mais concentrent 85% des mutations potentiellement responsables des maladies, des méthodes permettant de "capturer" l'ensemble des séquences codantes ("Whole Exome") et d'en déterminer la séquence ("Whole Exome Sequencing") ont été développées Choi M et al. , Genetic diagnosis by whole exome capture and massively parallel DNA sequencing Proc Natl Acad Sci U S A. 2009 Nov 10;106(45):19096-101. Epub 2009 Oct 27 Ces méthodes de "Whole Exome Sequencing" (WES) permettent d'identifier en théorie la quasi totalité des variations de séquences qui existent au niveau des séquences codantes entre des sujets atteints de la maladie multifactorielle et des sujets indemnes. On voit que cette approche WES est bien adaptée à la caractérisation des variants rares qui étaient un peu laissés pour compte par la méthode GWAS.
Choi M et al. , Genetic diagnosis by whole exome capture and massively parallel DNA sequencing Proc Natl Acad Sci U S A. 2009 Nov 10;106(45):19096-101. Epub 2009 Oct 27 Ces méthodes de "Whole Exome Sequencing" (WES) permettent d'identifier en théorie la quasi totalité des variations de séquences qui existent au niveau des séquences codantes entre des sujets atteints de la maladie multifactorielle et des sujets indemnes. On voit que cette approche WES est bien adaptée à la caractérisation des variants rares qui étaient un peu laissés pour compte par la méthode GWAS.


9/11






 Pritchard JK, Cox NJ, The allelic architecture of human disease genes: common disease-common variant...or not? Hum Mol Genet. 2002 Oct 1;11(20):2417-23
Pritchard JK, Cox NJ, The allelic architecture of human disease genes: common disease-common variant...or not? Hum Mol Genet. 2002 Oct 1;11(20):2417-23