2
-
Les banques de données utiles dans le domaine de la génétique
2
.
1
-
Les "Genome Browsers"
Ils correspondent à différentes bases de données qui permettent d’accéder aux données du génome humain (et de celui d’autres espèces) à l’aide d’une interface graphique. En plus des données de séquence, ces navigateurs permettent d’accéder à de nombreuses données d’annotation (gènes avec exons et introns, sites de fixation, régions d’homologie) (cf. 3.1).
Les plus populaires sont :
- Ensembl (European Bioinformatics Institute / Wellcome Trust Sanger Institute)
-
NCBI (National Cancer for Biology Information)
-
UCSC (University of California Santa Cruz)
D’autres méritent également le détour :
-
Vista (University of California)
-
Argo (BROAD Institute)
-
Mochiview (University of California Santa Cruz)
-
X :map (Paterson Institute for Cancer Research)
-
DiProGB (Leibniz Institute for Age Research)
-
Genatlas (Université René Descartes - Paris)
Si l’ensemble des "Genome Browsers" permet d’accéder à de très nombreuses données, aucun d’entre eux ne génère ces données. Ils sont donc dépendants d’autres centres ou laboratoires de recherche qui eux les produisent. Ceci explique pourquoi les mêmes données sont partagées par ces différents navigateurs et c’est souvent l’interface qui oriente vers l’un plutôt que l’autre ou la richesse des outils d’analyse associés.
Il existe cependant des "Genome Browsers" dédiés à un projet de recherche particulier. Dans ce cas, leur champ d’action est plus réduit mais ils fournissent
directement les données et sont donc responsables de leur qualité. Il est en effet critique de s’assurer de la qualité des données collectées dans une base de données car si elle est ouverte à tous, sa qualité ne pourra être assurée et les données qu’elle contient seront vite d’une utilité limitée comme nous le verrons dans le chapitre dédiée aux banques de données de mutations (cf. 2.5.1).
Trois bases de données illustrent bien cette catégorie :
-
James Watson’s Personal Genome Sequence (Baylor College of Medicine)
-
Craig Venter’s Personal Genome Sequence (Craig Venter Institute)
-
1000 genomes project (Projet international)
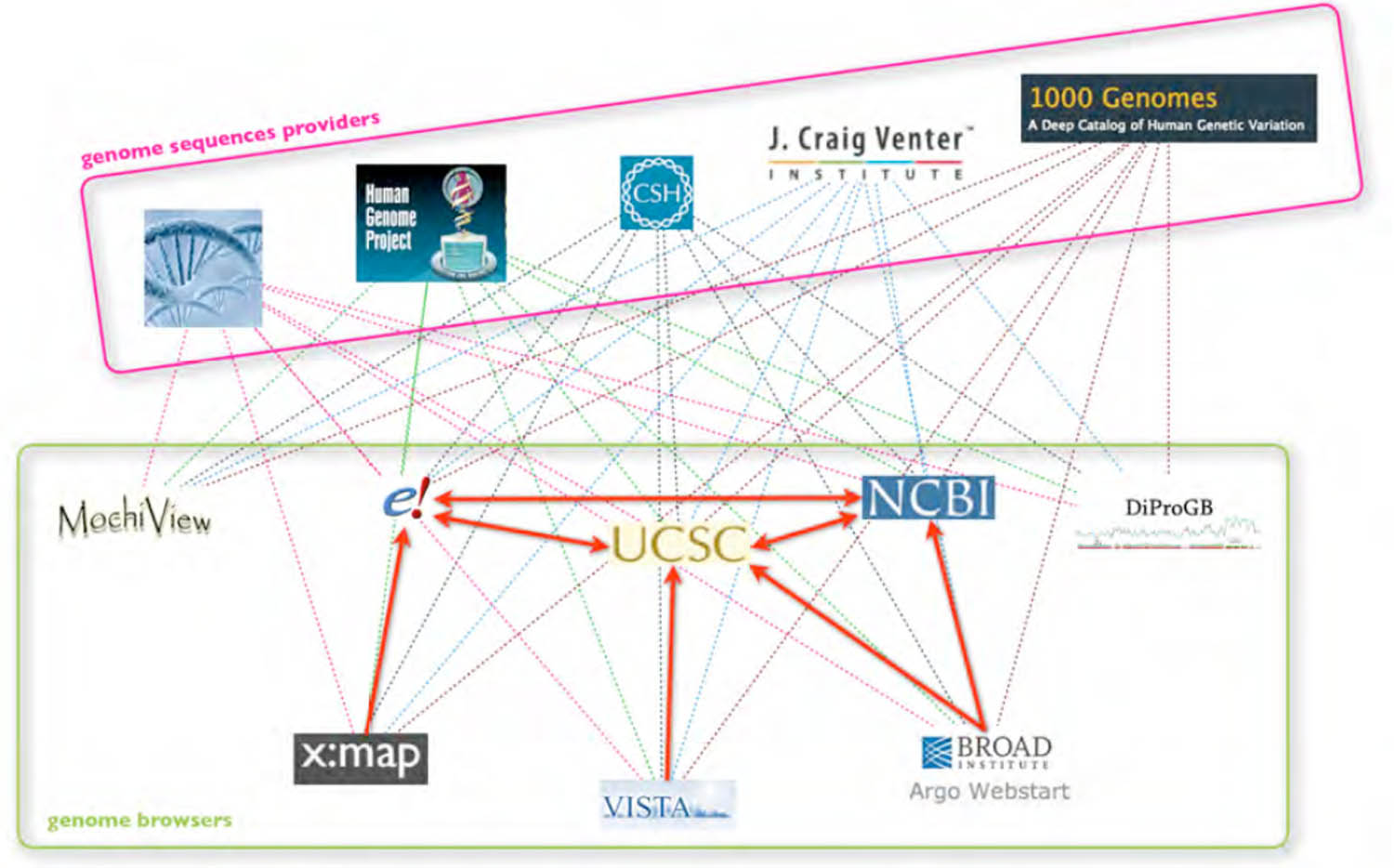
Comme nous l’avons vu, les différents "Genome Browsers" partagent des données brutes (séquence de référence) mais également des données d’annotation. Comme le montre la figure 1, il existe ainsi des relations complexes entre les fournisseurs de données et les "Genome Browsers".
Rectangle rose = fournisseurs de données : centres de séquençage académiques et privés, centres de séquençage et d’assemblage du projet génome humain, projets de séquençage de génomes personnels (James Watson, Craig Venter …), projet 1 000 génomes. Rectangle vert = Genome Browsers. Lignes pointillées = données utilisées par les génomes Browsers. Flèches rouges = liens entre les différents Genome Browsers.
2
.
2
-
L’annotation : outils et bases de données
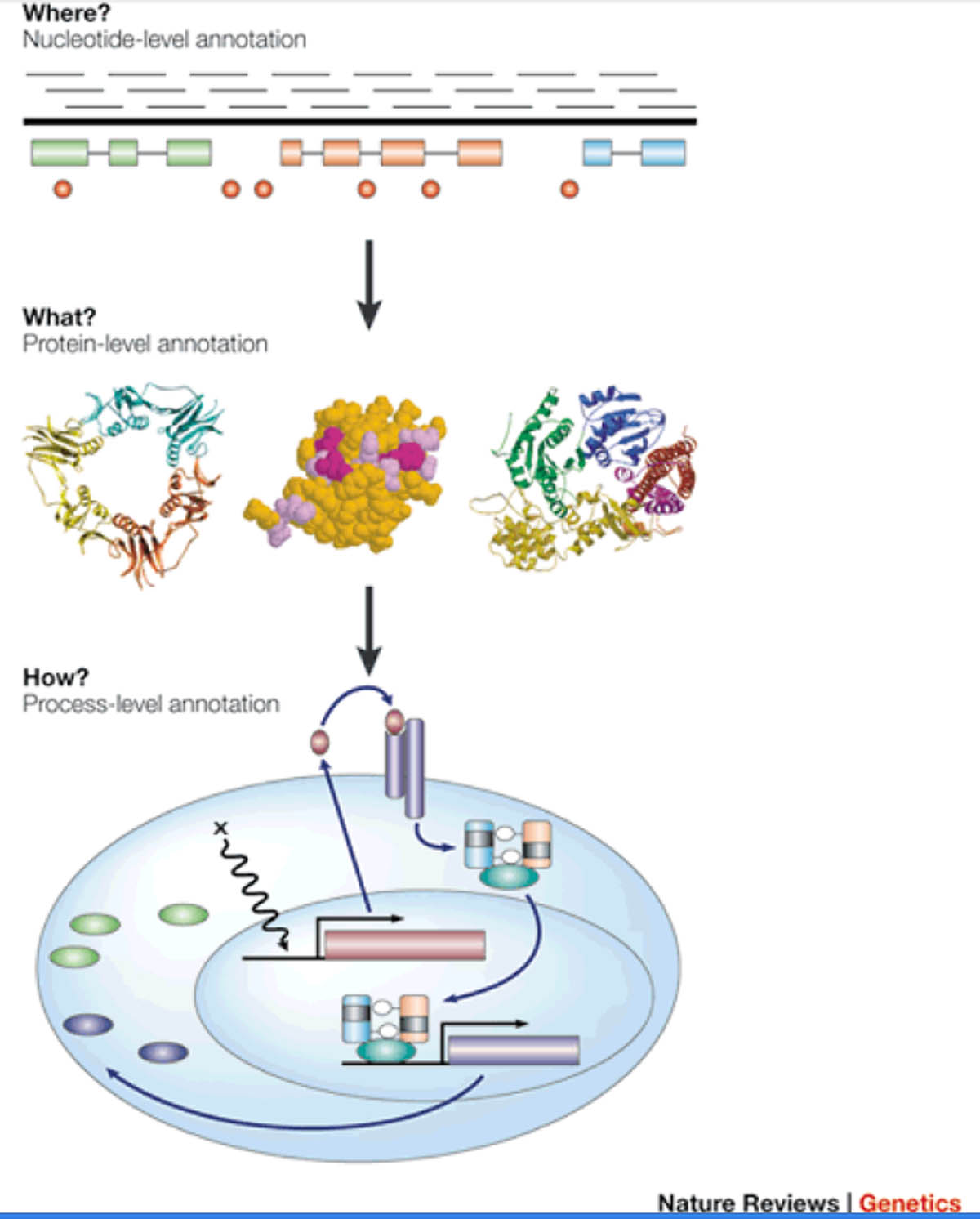
La connaissance de la séquence du génome humain n’aurait qu’une portée limitée si elle n’était annotée à différents niveaux. Ainsi l’annotation est un processus
complexe qui peut être subdivisé en trois catégories : l’annotation syntaxique, l’annotation fonctionnelle et l’annotation relationnelle (figure 2) :
L'annotation syntaxique qui permet d’identifier les séquences présentant une pertinence biologique (gènes, signaux, répétitions, …)
L'annotation fonctionnelle qui permet de prédire les fonctions et produits potentiels des gènes préalablement identifiés (similitudes de séquences, motifs,
structures, ...) et de collecter d'éventuelles informations expérimentales (littérature, jeux de données à grande échelle, …)
L'annotation relationnelle qui permet enfin de déterminer les interactions que les objets biologiques préalablement identifiés sont susceptibles d'entretenir (familles de gènes, réseaux de régulation, réseaux métaboliques, …).
2/6